6.1 결합(Append)
library(tidyverse)6.1.1 데이터 프레임의 결합(append)
종종 데이터 세트들은 여러 개의 파일로 분리되어 있는데, 이는 아마도 데이터들이 여러 원천에서 또는 여러 연구자들이 수집하기 때문일 것이다.
파일들이 같은 변수들을 공유하고 있다면(희망사항이긴 하지만), 데이터 세트들을 결합하거나 행들을 함께 묶을 수 있다.
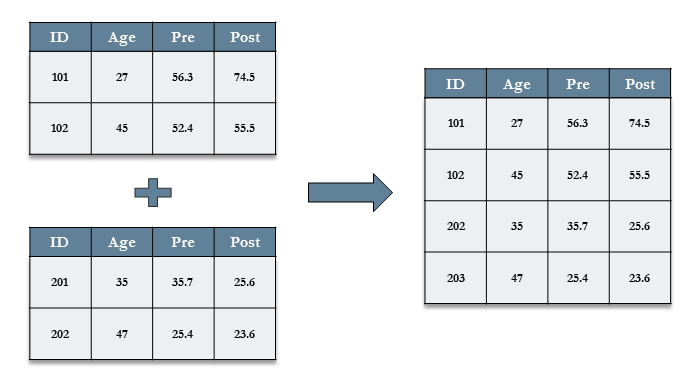
- 위의 그림에서 보듯이 결합(append)은 여러 개의 파일들이 컬럼 변수들을 같이 공유할 때 이 파일들의 행(rows)을 묶어 준다.
위의 그림에서 위의 표는 tb1, 아래의 표는 tb2 등으로 하여, rbind()로 결합을 한 tb3는 다음과 같다.
# two tibbles
tb1 <- tibble(ID = c("101", "102"),
Age = c(27, 45),
Pre = c(56.3, 52.4),
Post = c(74.5, 55.5))
tb2 <- tibble(ID = c("201", "202"),
Age = c(35, 47),
Pre = c(35.7, 25.4),
Post = c(25.6, 23.6))
# append using rbind()
tb3 <- rbind(tb1, tb2)
tb3## # A tibble: 4 x 4
## ID Age Pre Post
## <chr> <dbl> <dbl> <dbl>
## 1 101 27 56.3 74.5
## 2 102 45 52.4 55.5
## 3 201 35 35.7 25.6
## 4 202 47 25.4 23.6# append using bind_rows()
tb4 <- bind_rows(tb1, tb2)
tb4## # A tibble: 4 x 4
## ID Age Pre Post
## <chr> <dbl> <dbl> <dbl>
## 1 101 27 56.3 74.5
## 2 102 45 52.4 55.5
## 3 201 35 35.7 25.6
## 4 202 47 25.4 23.6- 여기서
tb1과tb2의 컬럼 구조는 동일하다.
6.1.2 결합의 두 가지 방법 : rbind() 함수와 bind_rows()함수
이러한 결합은 base 패키지의 rbind() 함수나 dplyr 패키지의 bind_rows() 함수로 수행할 수 있다.
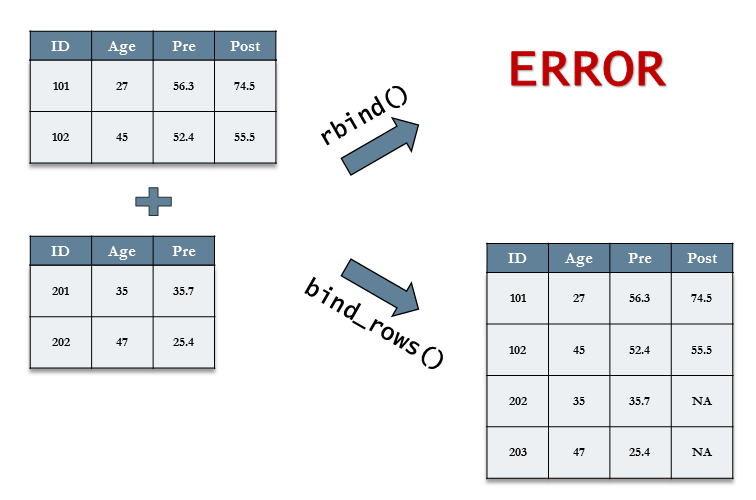
이 두 함수의 차이점은 대응되지 않는 컬럼이 있는 데이터 세트들을 결합할 때 처리방법이 다르다는 것이다.
rbind()함수는 에러가 발생한다.bind_rows()함수는 데이터 세트를 결합하되 대응되지 않는 컬럼에 대해서는 결측값인NA로 채우게 된다.
# two tibbles
tb1 <- tibble(ID = c("101", "102"),
Age = c(27, 45),
Pre = c(56.3, 52.4),
Post = c(74.5, 55.5))
tb2 <- tibble(ID = c("201", "202"),
Age = c(35, 47),
Pre = c(35.7, 25.4))
# append using rbind()
tb5 <- rbind(tb1, tb2)## Error in rbind(deparse.level, ...): numbers of columns of arguments do not matchtb5## Error in eval(expr, envir, enclos): 객체 'tb5'를 찾을 수 없습니다# append using bind_rows()
tb6 <- bind_rows(tb1, tb2)
tb6## # A tibble: 4 x 4
## ID Age Pre Post
## <chr> <dbl> <dbl> <dbl>
## 1 101 27 56.3 74.5
## 2 102 45 52.4 55.5
## 3 201 35 35.7 NA
## 4 202 47 25.4 NAtb1과tb2의 컬럼 구조가 서로 다른 경우rbind()를 이욯한 결합은 error가 발생bind_rows()를 이용한 결합의 경우에는 대응되지 않는 컬럼의 값은NA로 채워진다ㅏ.
이 두 함수들은 두 개의 데이터 세트에 있는 동일한 컬럼이 서로 다른 데이터 타입일 때 이를 처리하는 방법에도 차이가 있다. (예를 들어, 하나는 문자형이고 다른 하나는 숫자형인 경우)
rbind()함수는 강제적으로 형을 변환하여 결합한다.(강제 형 변환의 순서 : 논리형 > 정수형 > 더블형(실수형) > 문자형)bind_rows()함수는 에러를 발생시킨다.
# two tibbles
tb1 <- tibble(ID = c("101", "102"),
Age = c("27", "45"),
Pre = c(56.3, 52.4),
Post = c(74.5, 55.5))
tb2 <- tibble(ID = c("201", "202"),
Age = c(35, 47),
Pre = c(35.7, 25.4),
Post = c(25.6, 23.6))
# append using rbind()
tb7 <- rbind(tb1, tb2)
tb7## # A tibble: 4 x 4
## ID Age Pre Post
## <chr> <chr> <dbl> <dbl>
## 1 101 27 56.3 74.5
## 2 102 45 52.4 55.5
## 3 201 35 35.7 25.6
## 4 202 47 25.4 23.6# append using bind_rows()
tb8 <- bind_rows(tb1, tb2)## Error: Can't combine `..1$Age` <character> and `..2$Age` <double>.tb8## Error in eval(expr, envir, enclos): 객체 'tb8'를 찾을 수 없습니다tb1과tb2의 컬럼이 대응은 되지만 데이터 형이 다른 경우rbind()의 경우,tb7의Age컬럼이 문자형으로 변환됨bind_rows()의 경우, error 발생.
tb1과tb2의 컬럼이 대응이 되지 않는 경우는 모두 error 발생.
6.1.3 데이터 세트 결합의 예(대응되지 않는 컬럼이 있는 경우)
데이터 세트의 결합을 위해 2개의 데이터 세트 d1과 d2를 만들어 보자.
먼저 patient_pt1_dm.csv 파일을 불러와 d에 저장하고, mutate() 함수를 이용하여 agecat 변수와 highpain 변수(컬럼) 2개를 추가하여 d1을 만든다.
d2는 patient_pt1_dm.csv 파일을 불러와 저장한다.
이들 두 개의 데이터 세트들은 d1이 mutate() 함수에 의해 생성된 2 개의 컬럼 이외에는 다 같은 컬럼들을 가지고 있다.
# new data set that contains the same variables as d, except is missing 2 of them
d <- read_csv("data6/patient_pt1_dm.csv")##
## -- Column specification --------------------------------------------------------
## cols(
## .default = col_double(),
## hospital = col_character(),
## docid = col_character(),
## dis_date = col_character(),
## sex = col_character(),
## familyhx = col_character(),
## smokinghx = col_character(),
## cancerstage = col_character(),
## wbc = col_character()
## )
## i Use `spec()` for the full column specifications.d1 <- mutate(d,
agecat = cut(age, breaks=c(30,40,50,60,70,120)),
highpain = pain > mean(pain))
d2 <- read_csv("data6/patient_pt2_dm.csv")##
## -- Column specification --------------------------------------------------------
## cols(
## .default = col_double(),
## hospital = col_character(),
## docid = col_character(),
## dis_date = col_character(),
## sex = col_character(),
## familyhx = col_character(),
## smokinghx = col_character(),
## cancerstage = col_character(),
## wbc = col_character()
## )
## i Use `spec()` for the full column specifications.d1과 d2의 행과 열의 갯수 확인
# rows and columns of d2 and d1
dim(d1)## [1] 120 26dim(d2)## [1] 111 24이제 두 rbind() 함수와 bind_rows() 함수를 이용하여 두 데이터 세트를 결합해 보자.
rbind()함수를 이용하여 결합하면 에러가 날 것이다.bind_rows()함수는 첫 번째 데이터 세트에만 두 번째 데이터 세트의 관측치에 해당하는 부분을NA로 입력할 것이다.
가장 좋은 방법은 두 번쨰 데이터 세트인 d2에 대해서도 같은 변수들을 추가한 다음 결합함수를 이용하는 것이다.
그러나, bind_rows() 함수가 어떻게 작동하는가는 보여주기 위해, 우리는 현재의 상태로 데이터 세트들을 결합한다.
원천 데이터 세트를 식별해 주는 변수를 생성하기 위해 bind_rows() 함수의 인수로 .id = 를 사용한다.
6.1.3.1 bind_rows() 함수의 이용 예
데이터 세트 d1과 d2를 bind_rows() 함수로 결합하여 d3를 만들어 보자.
# a new variable called source is added to the beginning of the dataset
d3 <- bind_rows(d1, d2, .id="source")
dim(d3)## [1] 231 27# these are the rows where the datasets meet
# hospital is found in both datasets, agecat and highpain are not
select(d3, source, hospital, agecat, highpain)[118:123,]## # A tibble: 6 x 4
## source hospital agecat highpain
## <chr> <chr> <fct> <lgl>
## 1 1 UCSF (50,60] TRUE
## 2 1 UCSF (50,60] TRUE
## 3 1 UCSF (50,60] TRUE
## # ... with 3 more rowsbind_rows()함수 내의.id = “source”에 의해d3에source컬럼이 추가되고, 그 값은d1에서 결합된 행인 경우(118 ~ 120 행)는1,d2에서 결합된 행이면(121~123 행)2의 값을 갖는다.d2에서 결합된 행의 경우agecat컬럼과highpain컬럼의 값이NA로 채워져 있음을 알 수 있다.
6.1.3.2 rbind() 함수의 이용 예
# this will work because we restrict d1 to only variables common to both
drbind <- rbind(d1[,1:24], d2)
drbind## # A tibble: 231 x 24
## hospital hospid docid dis_date sex age test1 test2 pain tumorsize co2
## <chr> <dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 UCLA 1 1-1 6-Sep-09 male 65.0 3.70 8.09 4 68.0 1.53
## 2 UCLA 1 1-1 7-Jan-11 fema~ 53.9 2.63 0.803 2 64.7 1.68
## 3 UCLA 1 1-1 4-Sep-10 male 41.4 -99 2.13 3 86.4 1.45
## # ... with 228 more rows, and 13 more variables: wound <dbl>, mobility <dbl>,
## # ntumors <dbl>, remission <dbl>, lungcapacity <dbl>, married <dbl>,
## # familyhx <chr>, smokinghx <chr>, cancerstage <chr>, lengthofstay <dbl>,
## # wbc <chr>, rbc <dbl>, bmi <dbl>rbind()함수를 이용하였으나 에러가 발생하지 않았다. 그 이유는d1의 컬럼을d2의 컬럼과 같게 만들기 위해d1[, 1:24]로 제한했기 때문이다.
# But this will not work
drbind1 <- rbind(d1, d2)## Error in rbind(deparse.level, ...): numbers of columns of arguments do not matchdrbind1## Error in eval(expr, envir, enclos): 객체 'drbind1'를 찾을 수 없습니다d1의 컬럼과d2의 컬럼이 일치하지 않기 때문에rbind()함수는 에러를 발생시키고 작업을 중단함을 알 수 있다.